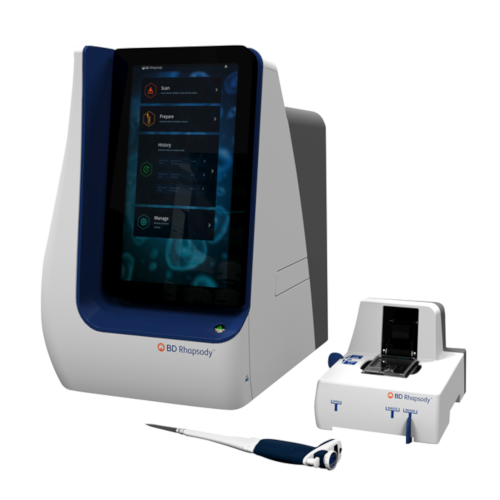
BD Rhapsody (BD Biosciences, США) – платформа для мультиомного анализа клеток, поддерживающая исследование транскриптома единичных клеток (scRNA-seq) и определение белковых клеточных маркеров. Для иммунологических исследований дополнительно обеспечивается анализ антиген-специфичных Т- и В-клеток, анализ VDJ рекомбинации. Система работает с клетками крови, клеточными культурами, препаратами клеток из нормальных и опухолевых тканей. В одном эксперименте возможен анализ от 100 до 60 000 индивидуальных клеток из одного или нескольких образцов с учетом сотен или тысяч характеристик. Результаты исследования используются для анализа гетерогенности клеточных популяций, для оценки ответа различных образцов на экспериментальные воздействия или терапию, для поиска связанных с клеточными процессами биомаркеров.
В линейку продуктов BD Rhapsody входят:
Только для исследовательских целей.
Принцип технологии основан на захвате мРНК и молекулярных меток из индивидуальных клеток баркодированными олигонуклеотидами, которые связаны с магнитными частицами. После сбора магнитных частиц выполняется синтез кДНК и подготовка библиотек для NGS.

Молекулярные баркоды включают:
Взаимодействие клеток и частиц происходит в микрофлуидном картридже BD Rhapsody, который имеет имеет формат микропланшета и содержит 200 000 микроячеек. Один картридж поддерживает анализ от 100 до 60 000 клеток в одном эксперименте. Образцы клеток можно пулировать до загрузки, при этом обеспечивается исследование от 1 до 12 первичных образцов в одном эксперименте.
Загрузка клеточной суспензии в картридж выполняется на станции BD Rhapsody Express. Буферный раствор для загрузки поддерживает жизнеспособность клеток. Клетки оседают в ячейки под действием силы тяжести и не подвергаются воздействию агрессивных физических и химических факторов, что позволяет сохранить жизнеспособность клеток и их свойства. Благодаря избыточному количеству микроячеек доля клеточных дублетов минимальна.
Вслед за клетками в картридж добавляются магнитные частицы с олигонуклеотидными баркодами. Частицы соответствуют размеру ячеек, поэтому в каждую ячейку попадает не более одной частицы, а избыток удаляется. В ячейках с клетками формируются пары из клеток и частиц.


Картридж имеет прозрачное дно для наблюдения за клетками и частицами после загрузки. Визуализация проводится на сканере BD Rhapsody, который в автоматическом режиме подсчитывает количество живых клеток, а затем оценивает распределение частиц в ячейках.



Оценка жизнеспособности клеток выполняется благодаря окрашиванию клеток в картридже кальцеином – красителем, который светится в живых клетках и регистрируется в режиме флуоресценции на сканере BD Rhapsody или на флуоресцентных микроскопах и имиджерах. Это позволяет оценить качество материала на этапе загрузки и принять решение о переходе к следующим этапам: синтезу кДНК и подготовке библиотек для NGS. Данные о количестве клеток в картридже используются для расчета количества циклов ПЦР при подготовке библиотек.
Контроль состояния клеток на этапе загрузки позволяет исключать сомнительные образцы на ранних этапах исследования и обеспечивает уверенность в результате еще до синтеза кДНК и подготовки библиотек.
Собранные в картридже клетки лизируются, молекулы мРНК выходят из клеток и сорбируются на частицах, находящихся в тех же ячейках. После захвата мРНК станция BD Rhapsody Express при помощи магнита переносит все частицы в одну пробирку, где выполняется синтез кДНК. На этом этапе происходит индексирование продуктов: молекулы кДНК, полученные на мРНК из одной клетки, приобретают один и тот же индекс, связанный с одной частицей.
Полученный продукт используется для синтеза библиотек, которые совместимы с секвенаторами Illumina. После первой реакции амплификации частицы с кДНК могут быть собраны и использованы повторно для исследования по другому протоколу. В случае избыточного количества клеток частицы можно аликвотировать, чтобы использовать только часть образца.

Протоколы подготовки библиотек поддерживают полнотранскриптомный анализ, таргетный анализ экcпрессии генов с использованием различных панелей, мультиомный анализ с оценкой фенотипа, анализ VDJ рекомбинации. В одном эксперименте возможно сочетание нескольких протоколов.

Секвенирование выполняется на приборах производства компании Illumina в режиме парных прочтений. Индексированные библиотеки BD Rhapsody поддерживают пулирование образцов. Выбор условий секвенирования выполняется в зависимости от количества клеток в эксперименте, схемы исследования и производительности секвенатора.
Для первичной обработки файлов fastq с результатами секвенирования пользователям системы BD Rhapsody предоставляется доступ к облачной программной платформе Seven Bridges, которая определяет клетки и связанные с ними транскрипты, маркеры, метки. Работа с сервисом Seven Bridges не требует специальных знаний в области биоинформатики.

Последующий анализ данных выполняется в программе SeqGek (FloJo, США)- специализированном приложении для анализа и визуализации результатов мультиомного анализа. Программа поддерживает определение клеточных субпопуляций, анализ их характеристик, статистическую обработку, визуализацию на графиках t-SNE и UMAP. SeqGek включает программные инструменты, соответствующие способам обработки цитометрических данных, поэтому является доступным и интуитивно понятным средством для анализа результатов.
Анализ результатов может также выполнятся другими методами бионформатики с использованием различных пакетов программного обеспечения.
Платформа BD Rhapsody позволяет на одном образце реализовать несколько исследований.
Например, поддерживается одновременный анализ транскриптома и фенотипа.
Таргетный подход обеспечивает успешное разделение субпопуляций для известных типов клеток, при этом для анализа достаточно небольшого количества прочтений на клетку. Поэтому затраты на секвенирование значительно сокращаются, а анализ упрощается.
Для подготовки таргетных библиотек используются наборы праймеров, которые содержат панели для нескольких сотен генов.
При помощи праймеров выполняется отбор транскриптов в ходе амплификации кДНК.

BD Biosciences предлагает несколько готовых таргетных панелей для исследования клеток иммунной системы человека и мыши, а также панель для исследований РМЖ. Готовые панели могут быть дополнены пользовательскими праймерами. Также доступны полностью кастомизированные панели до 600 генов.
Таргетная панель
BD Rhapsody Immune Response Panel Human
399 генов, включая:
CD-маркеры, хемокины и хемокиновые рецепторы, иммуноглобулины, тканеспецифичные маркеры, белки передачи сигнала в клетке, маркеры пролиферации и апоптоза, транскрипционные факторы, ферменты, молекулы клеточной адгезии

Для анализа фенотипа клеток разработаны антитела BD AbSeq, которые содержат олигонуклеотидные метки, кодирующие специфичность антител. Олигонуклеотид включает полиА-последовательность, поэтому после обработки клеток антителами и лизиса клеток олигонуклеотиды связываются с частицами BD Rhapsody.
После подготовки библиотек и секвенирования исследователь получает информацию, какие поверхностные маркеры присутствовали на клетках. Анализ может выполняться как в сочетании с определением транскриптома клеток, так и самостоятельно.
В отличие от флуоресцентных меток, количество комбинаций олигонуклеотидных меток не ограничено. Поэтому такой метод анализа фенотипа превосходит проточную цитометрию, позволяя определять десятки и сотни параметров на каждой клетке.
Линия BD AbSeq включает несколько сотен антител к клеточным маркерам человека и мыши. Антитела заранее протестированы и не требуют подбора разведений . Для упрощения многопараметровых исследований компания BD Biosciences разработала готовый реагент из 30 антител к маркерам клеток иммунной системы – BD AbSeq Immune Discovery Panel. Антитела мечены молекулярными баркодами, поставляются в виде лиофилизированной смеси.

Набор для мультиплексного анализа позволяет исследовать до 12 образцов в одном эксперименте, используя один картридж. Он содержит 12 антител, связывающие домены которых взаимодействуют со всеми клетками человека или мыши. С антителами конъюгированы олигонуклеотидные последовательности, которые включают метку Sample Tag, определяющую принадлежность к какому-либо образцу.
В эксперименте можно использовать все 12 меток или меньшее количество. Например, если проводится исследование 8 образцов, потребуется один картридж и 8 меток Sample Tag. Объединение материала от образцов происходит на этапе сбора частиц из картриджа. Для всех образцов будет подготовлена одна библиотека.

Кат №
Название
Количество
Реагенты для подготовки клеток
633781
Набор BD Human Single Cell Multiplexing Kit для мультиплексного анализа клеточных образцов, 12 антител с баркодами на 12 образцов по 1 млн клеток
На 12 образцов
633793
Набор BD Mouse Single Cell Multiplexing Kit для мультиплексного анализа клеточных образцов, 12 антител с баркодами на 12 образцов по 1 млн клеток
На 12 образцов
Реагенты для загрузки клеток и синтеза кДНК
633733
Набор картриджей BD Rhapsody Cartridge Kit
4 картриджа
633731
Набор реагентов BD Rhapsody Cartridge Reagent Kit для загрузки клеток, лизиса и захвата мРНК
4 эксперимента
633773
Набор реагентов BD Rhapsody cDNA Kit для синтеза кДНК на матрице РНК на частицах
4 эксперимента
Реагенты для подготовки библиотек
633801
Набора реагентов BD Rhapsody WTA Amplification Kit для подготовки библиотек для полнотранскриптомного анализа
4 эксперимента
633774
Набора реагентов BD Rhapsody Target Amplification Kit для подготовки библиотек для таргетного анализа транскриптома
4 эксперимента
633750
BD Rhapsody Immune Response Panel Hs, таргетная панель для анализа клеток иммунной системы
4 эксперимента
633751
BD Rhapsody T cell panel Hs, таргетная панель для анализа экспрессии генов в Т клетках
4 эксперимента
633752
BD Rhapsody Onco-BC Panel Hs, таргетная панель для анализа экспрессии генов в клетках РМЖ
4 эксперимента
1. Транскриптомика одиночных клеток выявляет изменения мононуклеарных клеток периферической крови при сепсисе.
Miaoyun Wen et al. Single-cell transcriptomics reveals the alteration of peripheral blood mononuclear cells driven by sepsis. Ann Transl Med. 2020 Feb; 8(4): 125. doi: 10.21037/atm.2020.02.35
2. Массивно-параллельные подходы в single cell на основе белков и секвенирования для профилирования экспрессии генов.
Xuhuai Ji, Igor Goncharov et al. Protein- and Sequencing-based Massively Parallel Single-cell Approaches to Gene Expression Profiling. Bio-101, 2019 Feb; DOI:10.21769/BioProtoc.3161
3. Single cell секвенирование глиобластомы показывает восприимчивость центральной нервной системы.
Bingshan Wu, Weihong Wang et al. Single-Cell Sequencing of Glioblastoma Reveals Central Nervous System Susceptibility. Front. Oncol., 16 November 2020 / https://doi.org/10.3389/fonc.2020.566599
4. Таргетный Multi-omic анализ измеряет экспрессию белка и транскриптов с низким содержанием на уровне отдельных клеток.
Florian Mair, Jami R.Erickson et al. A Targeted Multi-omic Analysis Approach Measures Protein Expression and Low-Abundance Transcripts on the Single-Cell Level. Cell reports, V 31, 7 April 2020, / https://doi.org/10.1016/j.celrep.2020.03.063
5. Измененные субпопуляции моноцитов при болезни Кавасаки, выявленные с помощью одноклеточного секвенирования РНК.
Zhimin Geng, Yijing Tao et al. Altered Monocyte Subsets in Kawasaki Disease Revealed by Single-cell RNA-Sequencing/ Journal of Inflammation Research 221:14.
6. scRNA секвенирование демонстрирует внутриопухолевую гетерогенность папиллярной карциномы щитовидной железы.
Zhengshi Wang, Youlutuziayi Rixiati et al. Single-cell RNA sequencing demonstrates the intratumoral heterogeneityof papillary thyroid carcinoma/ doi: https://doi.org/10.1101/2021.02.24.21251881
Продолжая просмотр этого сайта, Вы соглашаетесь на обработку файлов cookie.
ООО «БиоЛайн» использует cookie-файлы для обеспечения стабильной работы нашего сайта и улучшения взаимодействия с пользователями. Для получения дополнительной информации вы можете ознакомиться с принципами и правилами использования cookie-файлов, которые содержатся в Положении об обработке персональных данных